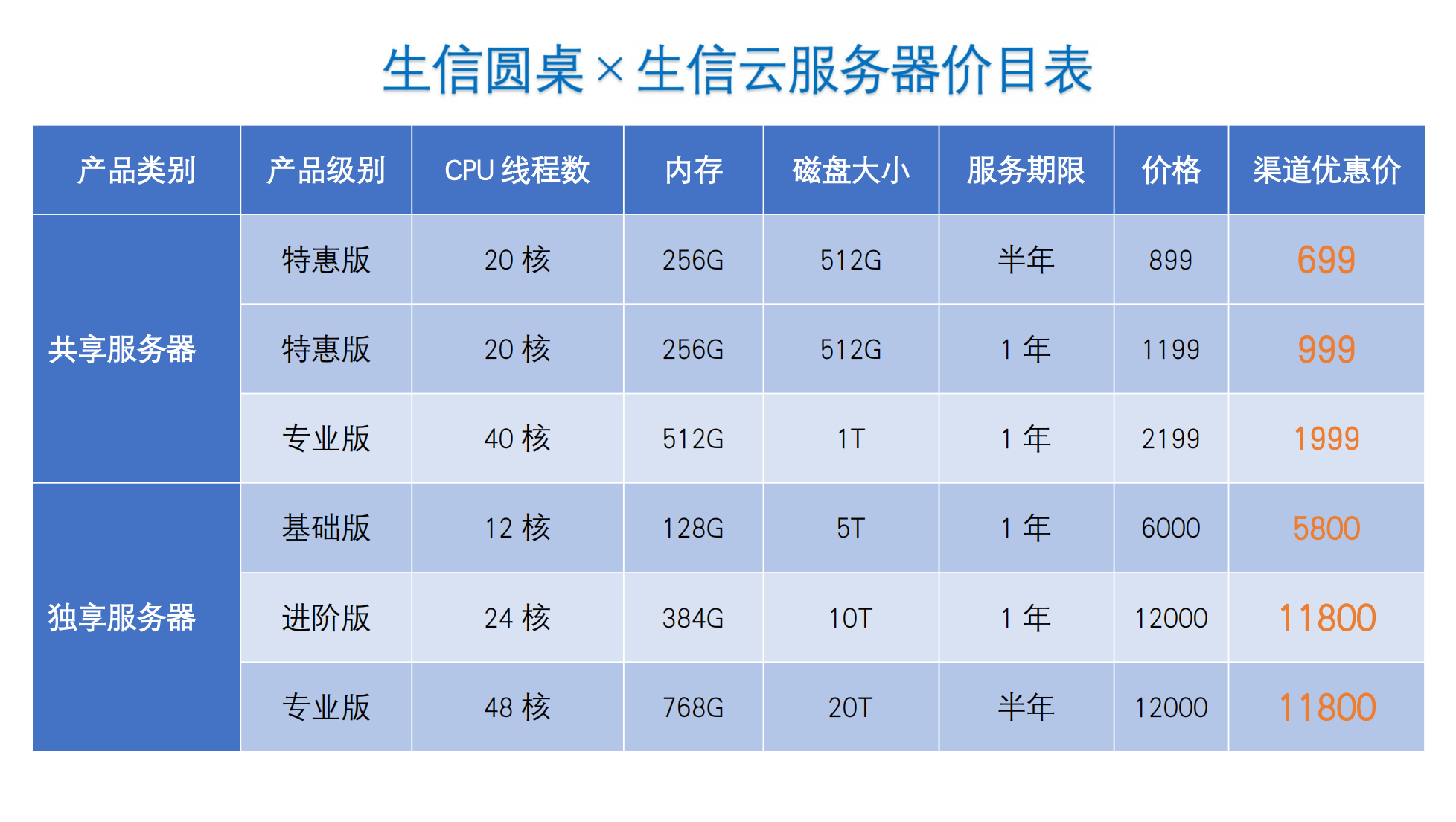
在单细胞生物信息学中,数据标准化和降维分析是理解和解释复杂数据的基础步骤。由于单细胞RNA-seq技术产生的数据具有高维度和稀疏性,数据标准化和降维处理成为了必须的操作,有助于消除实验间的技术性差异、提高分析的准确性,并揭示细胞间的潜在差异。
让生信分析更简单高效,点击图片访问【生信圆桌】获取生信云服务器
数据标准化方法:
单细胞RNA-seq数据通常会受到技术性因素的影响,例如测序深度、细胞捕获效率等。为了消除这些差异,研究人员需要对数据进行标准化处理,以便更公平地比较不同样本或细胞群体间的表达模式。常见的标准化方法包括:
1.总计数标准化(TPM、RPKM):
总计数标准化方法通过对每个基因的原始计数进行调整,使得所有样本的基因表达值在相同的尺度下进行比较。TPM(Transcripts Per Million)和RPKM(Reads Per Kilobase of exon model per Million mapped reads)是常见的两种总计数标准化方法。TPM通过对每个基因的读数进行标准化,使其能够反映基因在样本中的相对表达量,而RPKM则考虑了基因的长度和测序深度。
2.对数变换标准化:
对数变换是一种常用的标准化手段,通过对基因表达值进行对数转换(如log2转化),可以减小表达量差异的影响,并使数据更接近正态分布,适合后续的统计分析。对数变换可以减少高表达基因对模型的影响,提高对低表达基因的检测能力。
3.基于模型的标准化(SCTransform):
SCTransform是一种更加先进的标准化方法,它基于负二项分布模型,通过去除技术性噪声来标准化单细胞数据。该方法能够有效提高数据的质量,并且可以对低表达基因进行有效建模,减少技术噪音的影响。
通过这些标准化方法,我们能够最大程度地减小技术偏差的影响,使得数据中的生物学差异更加突出,从而提高后续分析的可信度。
降维技术介绍与对比:
降维技术是单细胞数据分析中的关键步骤之一,它帮助研究人员从高维数据中提取出潜在的结构和模式,使得数据可视化更加直观。常见的降维技术包括PCA、t-SNE和UMAP等,每种方法都有其优缺点。
1. 主成分分析(PCA):
PCA是一种经典的线性降维方法,它通过计算数据的协方差矩阵,找到最大方差的方向,将高维数据映射到低维空间。PCA的主要优点在于其数学原理简单且计算高效,能够快速减少数据的维度,帮助发现数据的主要成分。
优点:
计算速度快,适合处理大规模数据。
可解释性强,可以清楚地理解每个主成分代表的生物学意义。
对线性关系的数据效果很好。
缺点:
只能捕捉线性关系,对于复杂的非线性结构难以处理。
可能会丢失局部结构信息。
2. t-SNE(t分布随机邻域嵌入):
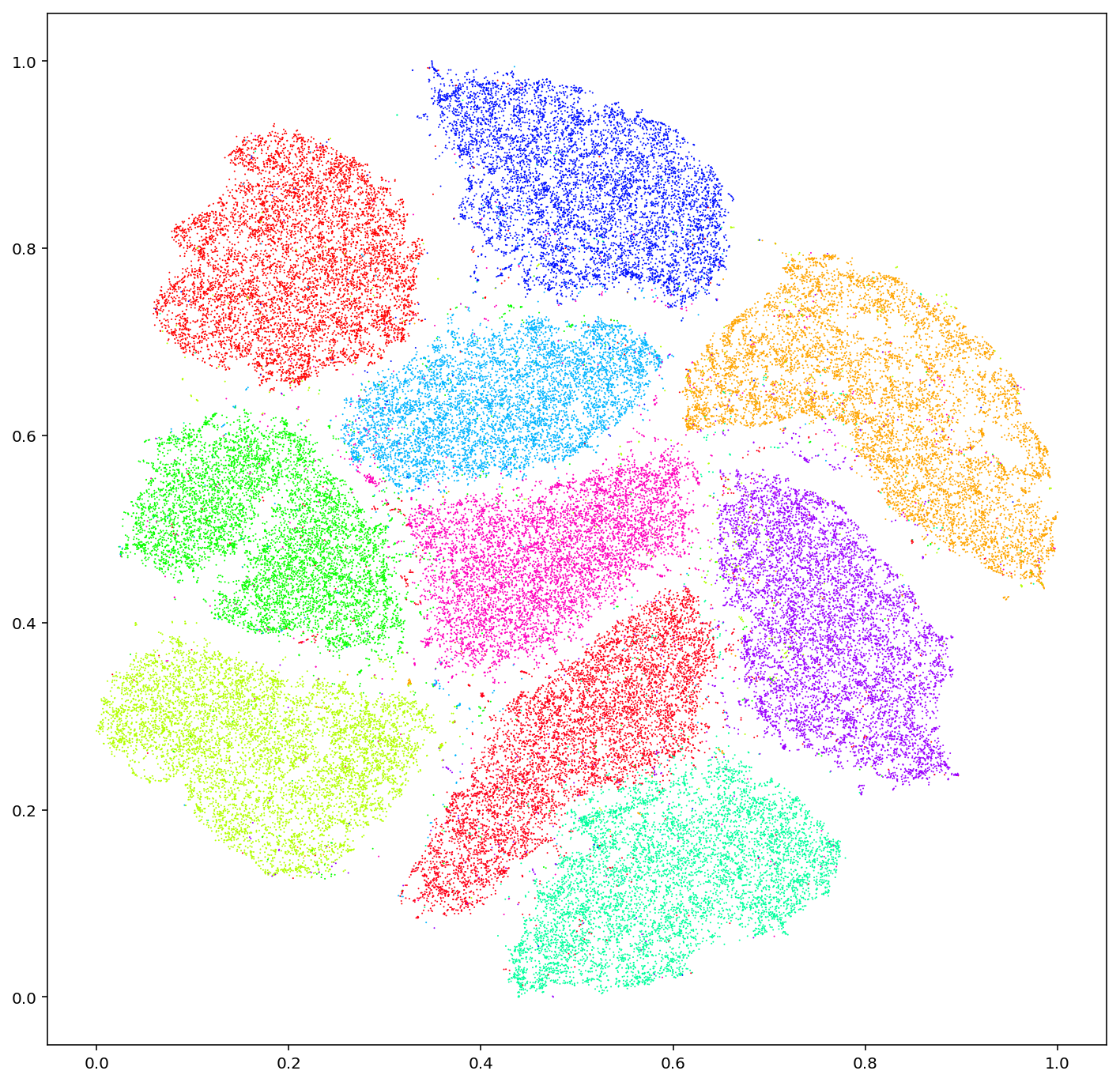
t-SNE是一种非线性的降维技术,特别适用于高维数据的可视化。它通过保持数据点在高维空间中的相对距离来进行降维,能够有效揭示数据中的局部结构。
优点:
非线性降维方法,能够捕捉数据中的复杂模式。
对细胞群体之间的差异表现出较好的分辨能力,特别适合用于聚类分析。
缺点:
计算开销较大,处理大规模数据时效率较低。
低维空间的坐标缺乏明显的生物学解释,难以进一步解读。
3. UMAP(统一流形近似与投影):
UMAP是一种近年来广受欢迎的降维技术,它结合了PCA和t-SNE的优点,能够在保持局部和全局结构的同时提供较好的计算效率。
优点:
兼具全局结构和局部结构的保留,能够更好地反映数据的整体趋势。
计算效率较高,适合处理大规模数据。
与t-SNE相比,UMAP的结果更具可解释性。
缺点:
参数较多,调参较为复杂。
结果的稳定性有时受到数据分布的影响。
降维方法的选择:
在单细胞数据分析中,选择合适的降维方法取决于数据的特点和分析目标。如果目的是进行初步的可视化,t-SNE和UMAP通常是更合适的选择,因为它们能够揭示复杂的细胞亚群结构。对于探索基因表达的主要变异成分,PCA是一个非常好的起点,尤其是在数据预处理阶段,常与其他降维方法结合使用。
总结:
单细胞数据的标准化与降维分析是高效解读生物学现象的核心步骤。通过合理的标准化方法,我们能够消除技术性噪音,使得数据更加可比。而降维分析则有助于揭示数据的潜在结构,发现细胞群体之间的差异。PCA、t-SNE和UMAP各有优缺点,在实际应用中,研究人员应根据分析需求和数据特性灵活选择合适的技术。